Résumé graphique. Crédit: Rapports de cellules (2024). DOI : 10.1016/j.celrep.2024.114967
Une équipe de recherche a récemment publié son article intitulé « CRISPRi-seq poolé de conception rationnelle découvre un inhibiteur de la peptidyl-ARNt hydrolase bactérienne » dans Rapports de cellules. Dans cette étude, l’équipe a créé une collection de mutants bactériens pour les aider à comprendre comment un nouveau type de molécule antimicrobienne, découverte grâce à leurs outils d’intelligence artificielle, empêche la croissance des bactéries.
Leurs recherches ont désormais permis d’identifier une combinaison unique d’un composé et de sa cible bactérienne, ouvrant ainsi des possibilités passionnantes pour le développement d’un nouvel antibiotique. L’équipe comprend le Dr ASM Zisanur Rahman, Julieta Novomisky Nechcoff et le Dr Silvia T. Cardona. Dans cette entrevue, l’Université du Manitoba s’entretient avec Cardona, professeure et directrice associée au Département de microbiologie et experte en découverte d’antibiotiques, pour fournir une compréhension plus approfondie de ses recherches et de ses travaux récents.
Nous avons entendu des inquiétudes concernant la montée de la résistance aux antibiotiques. Comment vos recherches y répondent-elles ?
Le défi de la résistance aux antibiotiques est crucial et de nouvelles approches pour découvrir les antibiotiques sont désespérément nécessaires. Si jamais vous vous demandez comment les scientifiques recherchent de nouveaux antibiotiques, vous savez peut-être qu’un travail de détective sérieux est nécessaire pour découvrir et comprendre comment les nouveaux composés tuent les bactéries infectieuses.
Pouvez-vous nous expliquer comment les antibiotiques tuent les bactéries ?
Oui. Un antibiotique tue les bactéries en se liant à une partie de leur machinerie cellulaire et en la perturbant. La machinerie cible exécute généralement un processus essentiel à la survie. Ainsi, lorsque l’antibiotique inhibe cette machinerie, la cellule meurt.
Comment avez-vous appliqué ce concept à votre recherche ?
La réduction de l’abondance de la cible d’un antibiotique peut rendre une cellule hypersensible à l’antibiotique. Dr Zisanur Rahman, un récent doctorat. diplômé dans mon laboratoire, j’ai appliqué une nouvelle technique intelligente utilisant les outils CRISPR pour identifier la cible des antibiotiques en réduisant l’abondance de différentes machines cellulaires et en recherchant des bactéries hypersensibles. Il s’agissait d’un travail considérable qui a nécessité une coordination avec l’étudiant à la maîtrise Jules Novomisky Nechcoff et le stagiaire de Mitacs Archit Devarajan.
Vous avez mentionné CRISPR, l’outil d’édition génétique. Qu’est-ce qui différencie votre outil CRISPR ?
Nous utilisons l’interférence CRISPR (également connue sous le nom de CRISPRi) au lieu du CRISPR classique, car cette technique ne coupe pas l’ADN, mais réduit plutôt l’expression d’un gène. Si nous considérons CRISPR classique comme un bouton muet, CRISPRi revient à baisser le volume. Cela nous permet d’étudier des gènes essentiels sans tuer la cellule que nous souhaitons analyser.
Pourquoi se concentrer sur les gènes essentiels ?
Parce que cibler des gènes essentiels est une bonne stratégie pour développer de nouveaux antibiotiques. Si vous interrompez une fonction cellulaire critique, les bactéries ne peuvent pas survivre. C’est logique, non ? Les frapper là où ça fait mal.
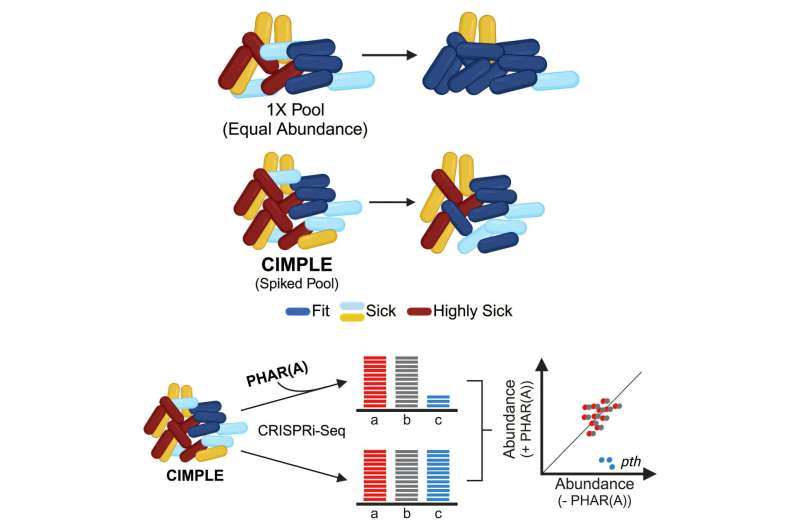
Pouvez-vous expliquer comment vous avez appliqué la conception rationnelle pour créer une bibliothèque mutante ?
À coup sûr. Les bibliothèques regroupées traditionnelles souffrent d’une croissance inégale des mutants, entraînant la perte de cibles importantes. Zisan a construit une bibliothèque super efficace de mutants CRISPR appelée CIMPLE, ce qui signifie : « bibliothèque regroupée de gènes essentiels médiée par CRISPRi ».
C’est une manière élégante de dire une collection de bactéries, chacune avec un gène différent détruit. En analysant soigneusement les caractéristiques de croissance de chaque mutant CRISPRi, nous pourrions prédire quels mutants seraient sous-représentés dans un système regroupé. Nous avons utilisé CIMPLE sur un tout nouvel inhibiteur de croissance, précédemment découvert par l’IA, un médicament mystérieux.
Découvrez les dernières nouveautés en matière de science, de technologie et d’espace avec plus de 100 000 abonnés qui comptent sur Phys.org pour des informations quotidiennes. Inscrivez-vous à notre newsletter gratuite et recevez des mises à jour sur les percées, les innovations et les recherches qui comptent :quotidiennement ou hebdomadairement.
Et comment avez-vous appliqué votre nouvel outil à la découverte d’antibiotiques ?
Nous avons utilisé CIMPLE sur un tout nouveau inhibiteur de croissance non caractérisé, un médicament mystérieux. Nous avons constaté que le nouveau composé cible une peptidyl-ARNt hydrolase, ou Pth, qui est une enzyme bactérienne essentielle qui contribue à la synthèse des protéines. Pour poursuivre les travaux sur cette cible, nous avons collaboré avec un expert de ces types d’enzymes : le Dr Yury Polikanov, de l’Université de l’Illinois à Chicago.
Quelle est l’importance de cette recherche pour le domaine de la découverte des antibiotiques ?
Le développement de CIMPLE fournit une méthode plus robuste et plus efficace pour identifier de nouvelles cibles antibactériennes, ce qui est crucial compte tenu du besoin urgent de nouveaux antibiotiques pour lutter contre la résistance aux médicaments. La découverte du Pth comme cible d’un nouvel antimicrobien est le résultat direct de cette méthodologie améliorée. Ainsi, cet article présente essentiellement un nouvel outil puissant pour la découverte d’antibiotiques et identifie une nouvelle cible pour les composés antibactériens.
Plus d’informations :
ASM Zisanur Rahman et al, CRISPRi-seq groupé de conception rationnelle découvre un inhibiteur de la peptidyl-ARNt hydrolase bactérienne, Rapports de cellules (2024). DOI : 10.1016/j.celrep.2024.114967
Fourni par l’Université du Manitoba
Citation: Q&A : Discovery ouvre des possibilités pour un nouvel antibiotique développé par l’IA (11 décembre 2024) récupéré le 11 décembre 2024 sur https://phys.org/news/2024-12-qa-discovery-possibilities-ai-antibiotic.html
Ce document est soumis au droit d’auteur. En dehors de toute utilisation équitable à des fins d’étude ou de recherche privée, aucune partie ne peut être reproduite sans autorisation écrite. Le contenu est fourni à titre informatif uniquement.